论文:草鱼D-loop多态性与幼苗生长性状的关联分析
草鱼D-loop多态性与幼苗生长性状的关联分析
傅建军1,2, 张 猛1, 沈玉帮1, 陈 勇3, 李家乐1
(1.上海海洋大学,省部共建水产种质资源发掘与利用教育部重点实验室,上海 201306;2.中国水产科学研究院淡水渔业研究中心,农业部淡水渔业与种质资源利用重点实验室,无锡 214081;3.通威股份有限公司水产工程技术研究中心,成都 610093)
为了探索草鱼(Ctenopharyngodonidella)线粒体DNA(mitochondrial DNA, mtDNA)多态性对生长性状的影响,鉴于mtDNA的母性遗传特征,本研究基于2011年繁殖用的20尾母本的D-loop序列信息,与通过亲子鉴定获得的853尾40日龄子代的体长、体质量进行关联分析。结果显示,草鱼6种D-loop单倍型对生长性状表型差异具有极显著影响(P<0.01);其中,单倍型为Hap16的子代的体长最大,并显著大于单倍型为Hap4的子代的体长(P<0.05);单倍型为Hap18和Hap16的子代的体质量较大,依次大于其他单倍型子代的体质量,并显著大于单倍型为Hap4的子代的体质量(P<0.05)。此外,草鱼D-loop序列各变异位点基因型对生长性状的影响水平不同;其中,Site01、Site06和Site07等3个位点对体长的差异存在显著影响(P<0.05),Site06和Site07等2个位点对体质量的差异存在显著影响(P<0.05)。研究表明,草鱼D-loop序列变异对子代生长性状具有显著影响,推测在草鱼生长性状改良的选育进程中,可以利用mtDNA多态性信息进行辅助选择。
草鱼(Ctenopharyngodonidella);D-loop序列;多态性;生长性状;关联性
草鱼(Ctenopharyngodonidella)是我国重要的大宗淡水鱼养殖品种之一,其养殖产量在世界淡水渔业中排名前列[1]。目前,选育生长优势的草鱼新品种,是满足日益增长的市场需求及实现其可持续利用的有效途径之一。然而,草鱼亲本个体大、成熟年限长,其常规选育进展较慢。通过筛选和利用与草鱼生长性状相关的分子标记,可以有效提高选择效率、缩短育种年限,对加快草鱼生长性状的改良进程具有重要意义[2]。
目前,研究人员主要从核基因组水平筛选与表型性状存在关联性分子标记,如SSR (Simple sequence repeats)标记[2],及草鱼谷胱甘肽硫转移酶[3]、醛缩酶A[4]、柠檬合酶[5]、PRL[6]和MSTN-1[7]等功能基因中筛选到一些与生长性状相关的SNP (Single nucleotide polymorphism)位点,这可为草鱼分子辅助育种提供辅助标记。作为重要的细胞质遗传物质,线粒体DNA (mitochondrial DNA,mtDNA)对维持动物正常生命活动有重要意义。在鱼类研究中,Danzmann等[8]和Ferguson等[9]研究表明mtDNA变异对虹鳟(Oncorhynchusmykiss)生长性状存在显著影响。就草鱼而言,基于mtDNA序列变异对草鱼群体遗传变异研究已有较多开展[10-12],而基于mtDNA变异与表型性状间的关联分析还未见报道。该研究基于草鱼育种材料,利用已发表文献中母本mtDNA的D-loop序列多态性信息[12]与子代生长性状[13]进行关联分析,探讨草鱼mtDNA变异与生长表型的关联性,以期丰富草鱼生长性状遗传改良的潜在辅助标记。
1 材料与方法
1.1 实验材料
2011年5月,在江苏吴江国家级四大家鱼原种场(苏州市吴江水产养殖有限公司)开展草鱼繁育工作。繁殖用亲本来源于珠江水系的肇庆群体[12],并剪鳍固定于无水乙醇,4 ℃保存,用于后续DNA提取。
实验中,对50尾亲本(25尾雌鱼、25尾雄鱼)采取人工激素结合水流刺激进行催产,在约20 m3圆形水泥池中进行。人工授精过程,用干燥洁净注射器收集雄鱼精液,并于4~8 ℃冰盒避光暂存;然后对催产成熟雌鱼逐个挤卵,根据雌鱼卵量,随机与2~4尾雄鱼精液混合,每个交配单元用500 mL卵子与2 mL精液混合,于同一孵化桶(约1 m3容积)内孵化,经过2 d连续的流水孵化(22~24 ℃)。水花鱼苗通过土池(2 000~2 500 m2,池中心水深1 m左右)培育,饲养密度为150尾/m2,下塘前通过施有机肥培育开口饵料,下塘3 d后泼洒豆浆,并保持池水肥度,下塘7 d后开始投喂人工颗粒料。
2011年7月,随机收集864尾40日龄夏花草鱼苗,测量体长和体质量数据;测量工具分别为游标卡尺(精确到0.002 cm)和电子天平(精确到0.01 g)。同时,剪尾部组织固定于无水乙醇,4 ℃保存,用于后续DNA提取。
1.2 DNA提取及亲子鉴定
对收集的草鱼鳍条组织使用改良的高盐法[14]提取基因组DNA,并利用1%琼脂糖凝胶电泳检测完整性,经NanoDrop 2000紫外分光光度计检测纯度和浓度,并稀释至50 ng/μL,于-20 ℃冰箱保存备用。
实验鱼的系谱关系通过基于12个微卫星标记的亲子鉴定技术构建[15]。对人工繁殖的亲本和子代个体进行基因型分析,基于似然法采用Cervus 3.0软件[16]确定亲子关系,分析中采用95%的置信度,并排除多于3个位点错配的亲子关系。
1.3 亲子数据整理与关联分析
该研究母本D-loop多态性信息来源于傅建军等[12]的文献,母本D-loop区(898 bp)的单倍型和变异位点信息采用DnaSP 5.0软件[17]统计;为了减少不同养殖池塘的影响,数据来源为Fu等[13]报道的同一池塘(Pond 01)的 853尾获得准确系谱关系的40日龄草鱼的体长和体质量数据,鉴定后代来自20尾母本(5尾参加繁殖的母本未获得后代)。
利用SPSS 16.0软件[18]对各单倍型及变异位点基因型与对应子代的生长性状进行单因素方差分析(One-Way ANOVA),多重比较的显著性检验采用Duncan法(新复极差法)。并利用一般线性摸型(General linear model,GLM)分析,其数学模型为:
Yij=μ+Mi+eij
式中,Yij为第i单倍型(或变异位点基因型)内第j个体的某性状观察值,μ为某性状总体平均值,Mi为第i单倍型(或变异位点基因型)的效应值,eij为个体随机残差项。
2 结果
2.1 D-loop单倍型与生长性状的关联性
方差分析如表1所示,不同单倍型子代生长性状差异达到极显著水平(P<0.01)。不同单倍型子代生长性状的描述性统计及多重比较结果如表2所示,研究发现6个单倍型对应子代生长性状具有显著差异(P<0.05),被划分为2个一致性子集。其中,单倍型为Hap16的子代的体长最大,并显著大于单倍型为Hap4的子代的体长(P<0.05);单倍型为Hap18和Hap16的子代的体质量较大,依次大于其他单倍型子代的体质量,且显著大于单倍型为Hap4的子代的体质量(P<0.05)。
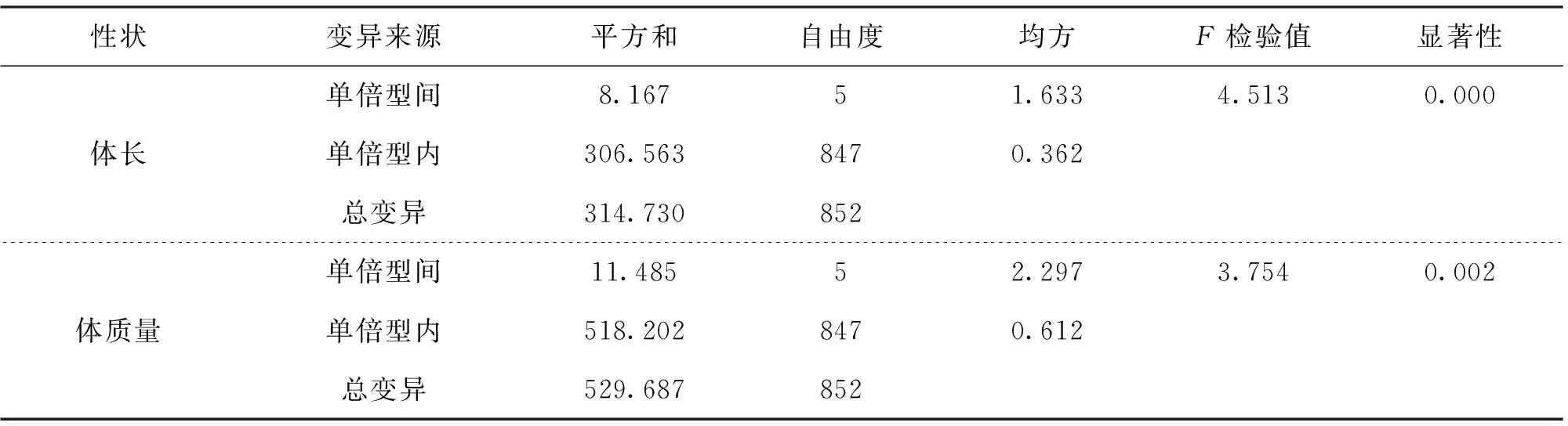
表1 草鱼不同D-loop单倍型子代生长性状的方差分析结果Tab.1 Variance analysis of offspring growth traits based on different D-loop haplotypes in C.idella

表2 草鱼不同D-loop单倍型子代生长性状的描述性统计及多重比较Tab.2 Descriptive statistics and multiplex comparisons of offspring growth traits based on different D-loop haplotypes in C.idella
注: 各性状同列多重比较,具有不同字母表示差异不显著(P<0.05)。
2.2 D-loop变异位点与生长性状的关联性
各变异位点基因型对应子代生长性状描述性统计与均值比较结果如表3所示,研究发现对体长有显著影响的变异位点有Site01(P<0.05)、Site06(P<0.01)和Site07(P<0.01);对体质量有显著影响的变异位点有Site06和Site07(P<0.05)。

表3 草鱼D-loop序列各变异位点基因型对应子代生长性状描述性统计和比较Tab.3 Descriptive statistics and comparisons of offspring growth traits based on different genotypes for each variable site of D-loop sequences in C.idella
3 讨论
mtDNA是重要的细胞质遗传物质,具有转录和翻译的功能,能合成与自身结构有关的一部分蛋白质。研究表明,mtDNA序列变异可能对动物经济性状造成不同程度影响。在家畜相关研究中,Sutarno等[19]研究发现牛(Bovine)mtDNA的D-loop区多态性与繁殖性能有显著关联;Jeon等[20]研究发现牛mtDNA 的COI、II、III基因变异与其重量性状差异存在显著相关;Biase等[21]研究发现牛mtDNA的tRNA基因变异与体质量及育种估值显著相关;Zhang等[22]研究发现牛mtDNA的ND5基因变异与早期生长性状差异有显著相关。在水产动物研究中,Danzmann等[8]和Ferguson等[9]研究发现虹鳟mtDNA变异对其生长性状存在显著影响;此外,Fisher等[23]研究发现贻贝属(Mytilus)mtDNA异质性与性别存在相关。
mtDNA作为母性遗传物质,该研究用母本mtDNA的序列替代对应子代的mtDNA信息,对于父本mtDNA不做考虑。研究发现草鱼不同D-loop单倍型及其变异位点基因型与子代生长性状差异存在显著的关联性。导致这种相关性的原因推测有以下2种可能:其一,线粒体作为能量代谢重要细胞器,其mtDNA变异可能引起能量代谢水平的差异,进而影响个体的生长表现;其二,mtDNA呈母系遗传,mtDNA多态性信息可能以母性遗传效应形式对后代生长表现造成影响。就该研究的实验材料而言,在Fu等[13]对40日龄草鱼生长性状的遗传参数估计中,发现母性效应对子代生长性状并不显著。考虑到该研究亲本(20尾母本)及个别单倍型子代数量(Hap16子代仅有4尾)相对较少,部分研究结果还有待扩充数量进一步验证;尽管如此,即便剔除子代数目较少的单倍型(Hap16)相应的数据,还能发现其他单倍型之间存在显著差异。因此,可以推测草鱼D-loop序列变异对子代早期生长性状存在影响,而mtDNA的功能基因多态性与生长表型的关联研究也值得期待。此外,相对于对所有个体的mtDNA测序或基因型检测,基于mtDNA的母系遗传特征,通过以母本mtDNA序列信息作为后代mtDNA序列信息的参考,有助于减少实验成本。可见,在草鱼生长性状的遗传改良和育种进程中,可尝试借助母本mtDNA多态性预测后代的生长表现,并为交配设计和选择提供辅助依据。
[1] FAO.The State of World Fisheries and Aquaculture 2012[M].Rome:FAO,2012: 36-40.
[2]王解香,白俊杰,于凌云.草鱼EST-SSRs标记的筛选及其与生长性状相关分析[J].淡水渔业,2012,42(1):3-8.
[3]刘小献,白俊杰,徐 磊,等.草鱼GSTR基因外显子1、外显子2的SNPs筛选及其与生长性状的关联分析[J].华中农业大学学报,2011,30(6):753-758.
[4]李玺洋,白俊杰,于凌云,等.草鱼醛缩酶A3-UTR突变与生长性状相关研究[J].淡水渔业,2012,42(5):13-16.
[5]樊佳佳,刘小献,白俊杰,等.草鱼柠檬酸合酶基因SNP筛选及与生长性状的关联分析[J].华中农业大学学报,2014,33(3):84-89.
[6]傅建军,张 猛,沈玉帮,等.草鱼PRL基因多态性与幼鱼生长性状和肌肉成分的关联分析[J].中国水产科学,2016,23(3):491-499.
[7]张 猛,陈 勇,沈玉帮,等.草鱼MSTN-1基因多态性与早期生长性状和肌肉成分关联分析[J].水产学报,2016,40(4):618-625.
[8] Danzmann R G,Ferguson M M.Heterogeneity in the body size of Ontario cultured rainbow trout with different mitochondrial DNA haplotypes[J].Aquaculture,1995,137(1-4): 231-244.
[9] Ferguson M M,Danzmann R G.Inter-strain differences in the association between mitochondrial DNA haplotype and growth in cultured Ontario rainbow trout (Oncorhynchusmykiss)[J].Aquaculture,1999,178(3-4): 245-252.
[10] 晏 勇,张兴忠,龙 华.草鱼线粒体DNA限制性内切酶分析[J].淡水渔业,1994,24(3):30-31.
[11]李树华,陈大庆,段辛斌,等.基于线粒体DNA标记的长江中游草鱼亲本增殖放流的遗传效果评估[J].淡水渔业,2014,44(3):45-50.
[12]傅建军,王荣泉,沈玉帮,等.我国草鱼野生群体D-Loop序列遗传变异分析[J].水生生物学报,2015,39(2):349-357.
[13] Fu J,Shen Y,Xu X,et al.Genetic parameter estimates and genotype by environment interaction analyses for early growth traits in grass carp (Ctenopharyngodonidella)[J].Aquacult Int,2015,23(6): 1427-1441.
[14] Rivero E R,Neves A C,Silva-Valenzuela M G,et al.Simple salting-out method for DNA extraction from formalin-fixed,paraffin-embedded tissues[J].Pathol Res Pract,2006,202(7): 523-529.
[15] Fu J,Shen Y,Xu X,et al.Multiplex microsatellite PCR sets for parentage assignment of grass carp (Ctenopharyngodonidella).Aquaculture International,2013,21(6): 1195-1207.
[16] Kalinowski S T,Taper M L,Marshall T C.Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment[J].Mol Ecol,2007,16(5): 1099-1106.
[17] Rozas J,Sánchez-Delbarrio J C,Messeguer X,et al.DnaSP,DNA polymorphism analyses by the coalescent and other methods[J].Bioinformatics,2003,19(18): 2496-2497.
[18] Cleophas T J,Zwinderman A H.SPSS for Starters[M].Springer Netherlands,2010: 15-18.
[19] Sutarno,Cummins J M,Greeff J,et al.Mitochondrial DNA polymorphisms and fertility in beef cattle[J].Theriogenology,2002,57(6): 1603-1610.
[20] Jeon G J,Chung H Y,Choi J G,et al.Relationship between genetic variants of mitochondrial DNA and growth traits in Hanwoo cattle[J].Asian Australas J Anim Sci,2005,18(3): 301-307.
[21] Biase F H,Meirelles F V,Gunski R,et al.Mitochondrial DNA single nucleotide polymorphism associated with weight estimated breeding values in Nelore cattle (Bosindicus)[J].Genet Mol Biol,2008,30(4): 1058-1063.
[22] Zhang B,Chen H,Hua L,et al.Novel SNPs of the mtDNA ND5 gene and their associations with several growth traits in the Nanyang cattle breed[J].Biochem Genet,2008,46(5-6): 362-368.
[23] Fisher C,Skibinski D O F.Sex-biased mitochondrial DNA heteroplasmy in the marine musselMytilus[J].Proc R Soc London,1990,242(1305): 149-156.
声明:本站属公益性没有商业目的的网站,上列文章仅供个人学习参考。本站所发布文章为原创的均标注作者或来源,未经授权不得转载,许可转载的请注明出处。本站所载文章除原创外均来源于网络,如有未注明出处或标注错误或侵犯了您的合法权益,请及时联系我们!
欢迎关注本站(可搜索)"养鱼E线"微信公众帐号和微信视频号"养鱼一线"以及头条号"水花鱼@渔人刘文俊"!

