论文:长江中上游4个鲢群体遗传多样性分析
长江中上游4个鲢群体遗传多样性分析
陈会娟,刘明典,汪登强,董微微2,,吕 浩,陈大庆,李 云
(1.西南大学动物科技学院,重庆 400715;2.西南大学生命科学学院,重庆 400715; 3.中国水产科学研究院长江水产研究所,武汉 430223)
鲢(Hypophthalmichthysmolitrix)作为“四大家鱼”之一,分布于全国各大江河及附属水体,长江是我国鲢天然种质资源的重要产地[1]。由于鲢在我国水产养殖和水生态环境修复中具有重要的地位,其遗传多样性一直是人们关心的主要问题。近年来监测表明,鲢的资源量正处于衰退之中,长江中上游部分江段鲢在渔获物中已不成优势种[2],并且仔鱼数量呈下降趋势[3]。为了在补充长江鲢资源的同时进行生态修复,近年来中国在长江各省均开展了增殖放流活动[4],增殖放流活动在修复渔业资源的同时,也会给增殖水域野生群体的遗传多样性、种群结构以及生态系统的结构与功能带来诸多风险[5-6]。另外,鲢为江湖洄游型鱼类,葛洲坝和三峡大坝的建设阻断了长江上游和中游鲢群体的交流,可能会导致其遗传多样性和遗传结构的变化。近期一些研究表明,河流大坝建设对鱼类特别是洄游型鱼类的遗传多样性和遗传结构造成了一定的影响[7-8]。
从上世纪80年代以来,不同学者采用不同的分子标记开展了鲢群体遗传学研究[9-10],取得了不少成果。研究结果表明,我国三条主要河流鲢群体间并没有明显的遗传分化,但与国外引入群体已经有显著遗传分化[11]。目前的研究较少涉及长江上游群体,仅见王长忠等[12]和庞美霞等[13]采用微卫星标记对长江上游群体进行了研究。长江中游和上游均存在鲢重要的自然产卵场,是维持长江中游种质资源的最重要场所。当前,长江鲢群体受到过度捕捞、大坝阻隔、水体污染、大规模增殖放流等人类活动的影响不断加剧,研究长江中上游鲢群体当前的遗传背景,比较上、中游群体遗传结构以及近年来遗传多样性的变化,对长江鲢群体的资源保护和科学管理具有重要意义。本研究采用线粒体Cytb基因以及D-loop序列对长江中上游4个鲢群体开展研究,比较上中游群体的遗传分化及近年来长江群体遗传多样性变化情况,以评估人类活动对鲢种质资源的影响,并为制定种质资源保护措施提供科学依据。
1 材料与方法
1.1 样本采集及基因组DNA提取
本实验4个鲢群体共151尾样本采于2016年6-9月渔业资源调查期间,样本均为渔民捕获的长江鱼苗,其中长江上游重庆江津江段(JJ)42尾、长江中游湖北宜昌江段(YC)30尾、嘉鱼江段(JY)34尾和黄冈江段(HG)45尾(图1)。每尾样本剪取少量鳍条,用无水乙醇保存,带回实验室。
基因组DNA提取方法采用酚-氯仿法[14]。
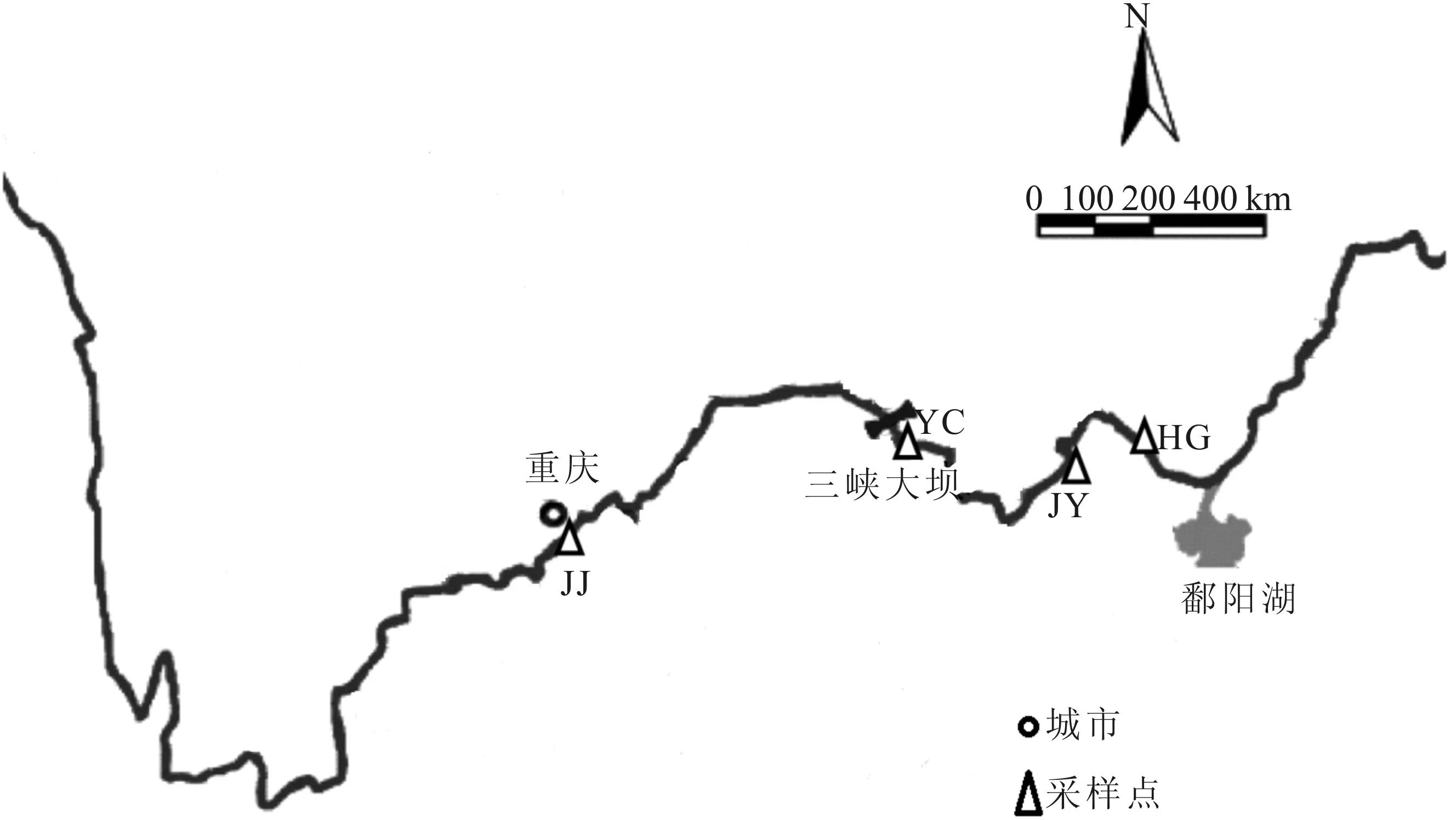
图1 长江中上游4个鲢野生群体的地理分布Fig.1 Location map of 4 populations of H.molitrix from middle and upper Yangtze River
1.2 PCR扩增及测序
分别扩增样本的线粒体DNA Cytb基因和控制区(D-loop)序列。
引物序列[15]:Cytb:L14724:5′-GACTTGAAAAACCACCGTTG-3′,H15915:5′-CTCCGATCTCCGGATTACAAGAC-3′;D-loop[16]:MitDI-F:5-CACCCYTRRCTCCCAAAGCYA-3′,MitDI-R:5′ -GGTGCGGRKACTTGCATGTRTAA-3′。
PCR 的 反 应 总 体 积 为 50 μL,其 中 包括 10×Buffer(Mg2+)5 μL、模板 45~60 ng、Taq 酶(5 U/L)0.4 μL、dNTPs(10 mmol/L)0.6 μL、10 μmol/mL上下引物各 2 μL,最后用灭菌双蒸馏水补至 50 μL。反应程序为:94 ℃预变性 5 min;94 ℃变性 30 s,55 ℃退火 30 s,72 ℃延伸 90 s,共35个循环;72 ℃延伸 6 min。PCR 产物检测选用1%的琼脂糖凝胶电泳,然后送往测序公司双向测序。
1.3 数据分析
测定的 DNA 序列经手工校对后,用ClustalX[17]对序列进行对位排列。利用MEGA 6.0[18]分析软件计算群体间遗传距离、统计序列的平均碱基组成、计算转换/颠换比率。DnaSP 5.0软件[19]被用来统计单倍型多样性(Hd)和核苷酸多样性(Pi)及种群间基因流情况(Nm);以单倍型为基础的网络结构图用Network 4.6.1.0软件构建。以Arlequin 3.11 软件中的方差分析(AMOVA)[20]方法估算遗传变异固定指数(FST)值,设置1 000次重复随机抽样重排后进行显著性检验。
2 结果与分析
2.1 序列变异分析
151个样本共扩增出135条Cytb序列,经比对后序列长968 bp。分析显示,转换数明显高于颠换数,Ts/Tv=10.39。A、T、C、G平均碱基组成为29.12%,29.28%,27.60%和14.00%,T+A含量(58.4%)高于G+C(41.6%),表现出明显的反G偏倚,显示鱼类Cytb基因的共同特征。共发现53个(5.5%)变异位点,包括44个(4.5%)简约信息位点和9个(0.9%)单变异位点。135个个体中,共定义了19个单倍型。
151个样本共扩增出150条D-loop序列,序列长890 bp,Ts/Tv=8.25。A、T、C、G平均碱基组成为33.65%,32.50%,20.11%和13.74%,同Cytb一样,表现出明显的反G偏倚。共发现94个(10.6%)变异位点,包括83个(9.3%)简约信息位点和11个(1.2%)单变异位点。150个个体中,共定义了48个单倍型。
2.2 群体遗传多样性分析
如表1 所示,Cytb序列中各群体的单倍型多样性介于0.416~0.810之间,核苷酸多样性介于0.001 71~0.015 30之间,其中江津(JJ)群体的单倍型多样性指数和核苷酸多样性指数均最高;D-loop序列中各群体的单倍型多样性介于0.874~0.918之间,核苷酸多样性介于0.007 17~0.029 23之间,同Cytb分析结果一样,江津(JJ)群体的单倍型多样性指数和核苷酸多样性指数均最高。

表1 鲢群体内遗传多样性Tab.1 Genetic diversity parameters within populations of H.molitrix
注:GN-群体名字;S-多态性位点数;Hd-单倍型多样性;Pi-核苷酸多样性;HN-单倍型数
2.3 群体间的遗传分化
4个群体分子方差分析(AMOVA)结果显示,2种标记的遗传分化指数(FST)均达到了极显著水平(FST=0.186 36**,Cytb标记;FST=0.083 94**,D-loop标记),表明4个群体间出现了遗传分化。对4个群体进行两两分析,FST和基因流如表2所示,不管是Cytb序列或是D-loop序列,就FST来说,JJ群体与中游3个群体均出现了显著的遗传分化,中游3个群体之间无分化;就基因流来说,江津群体与另外3个群体之间并不是随机交配的,存在遗传分化现象(Nm绝对值小于4),中游3个群体之间基因交流频繁,无遗传分化现象(Nm绝对值均明显大于4)。

表2 鲢4个群体间的遗传分化指数(对角线上) 和基因流(对角线下) Tab.2 Pairwise gene flow(below diagonal)and genetic differentiation index Fst(above diagonal) among 4 populations of H.molitrix
注:*说明遗传分化显著(P<0.05);**说明遗传分化极显著(P<0.01)
2.4 群体的单倍型进化关系
Cytb序列定义的19个单倍型如图2 A。其中H_1分布于4个群体,且分布最为广泛,频率最高,可能是原始单倍型。19个单倍型中,共享单倍型有7个,比例较低(36.8%),其中有3个为4个群体所共有。
D-loop序列定义的48个单倍型如图2 B。单倍型分布比较分散,没有明显的中心,其中Hap_2分布于4个群体,且分布最为广泛,可能是原始单倍型。除了Hap_2频率较高以外(20.5%),其他单倍型频率均较低,有18个单倍型为2个及2个以上群体所共有,仅有2个单倍型是4个群体共有。

图2 鲢单倍型中值连接网络分析图(A:Cyt b;B:D-loop)Fig.2 The haplotype network of H.molitrix(A:Cyt b;B:D-loop) 每个单倍型按地理分布不同用不同的颜色表示;圆圈大小表示该单倍型出现的次数。
基于Kimura 2-parameter 模型用单倍型数据构建N-J(Neighbour-Joining)系统发育树,如图3所示,Cytb序列单倍型构建的N-J树明显分为2个谱系,H_9单独为一个谱系,其他单倍型分为一个谱系,2个谱系间平均K2P遗传距离为0.039 03,分化水平极显著(FST=0.935 61,P<0.01)。2个谱系内的平均核苷酸变异数(k)分别为2.583和0.000,2个谱系间为7.245;D-loop序列单倍型构建的N-J树也明显分为2个谱系,Hap_24/25/35/48为一个谱系,其他单倍型为一个谱系。2个谱系间平均K2P遗传距离为0.076 24,分化水平极显著(FST=0.885 77,P<0.01)。2个谱系内的平均核苷酸变异数(k)分别为7.226和2.133,2个谱系间为58.550。

图3 鲢基于单倍型构建的N-J系统发育树(A:Cyt b序列;B:D-loop序列)Fig.3 The N-J phylogenetic trees based on haplotype(A:Cyt b;B:D-loop)
3 讨论
本研究结果显示,长江中上游4个鲢群体遗传多样性高于赣江群体(D-loop:Hd=0.727,Pi=0.008 97,935 bp)而低于瑞昌群体(D-loop:Hd=0.978,Pi=0.011 35,935 bp)的研究结果[21],也低于日本利根川盆地的引进鲢群体(D-loop:Hd=0.920,Pi=0.011)[22],总体上处于中等水平。无论是Cytb还是D-loop序列都显示,江津群体遗传多样性都高于中游的群体,这与王长忠等[12]采用微卫星标记的研究结果是一致的,证实了长江上游鲢的群体遗传多样性高于中游群体。由于缺乏葛洲坝及三峡工程截流前的数据,长江上中游鲢群体的这种差异是种群历史演化形成的,还是人类活动造成的,还有待进一步研究。同时,分析发现,宜昌群体的遗传多样性是最低的,特别是Cytb遗传多样性水平明显低于其他群体(表1),由于采样是在一个时间段从不同渔民渔获物中随机采集,可以排除采样误差,考虑到2016年长江中游雨量较往年大,造成沿江养殖场养殖鱼类大量逃逸,而宜昌附近的清江高坝洲、隔河岩等水库泄洪,引起大量鱼类进入长江,这是否会降低该江段鲢遗传多样性水平,需进一步分析。
从遗传分化指数来看,江津群体与其余群体间遗传分化均达显著水平(P<0.05)或极显著水平(P<0.01),而长江中游3个群体间遗传FST均低于5%(表2),说明上游与中游群体发生了显著遗传分化,中游群体间没有发生遗传分化,从基因流数据看,江津群体与中游3个群体间基因流均在1与4之间(表2),说明江津群体与中游群体间基因交流受到一定阻碍,这与王长忠[12]等运用微卫星分子标记的研究结果一致。从单倍型上看,频率最高的单倍型(Cytb:H_1,D-loop:Hap_2)为所有群体共享,但江津群体中频率第二的单倍型(Cytb:H_9,D-loop:Hap_24),只有一尾来自宜昌群体共享于H_9,同时中游群体中频率第二的单倍型(Cytb:H_2,D-loop:Hap_7)也只有一尾来自江津群体与之共享(图2)。比对试验样本发现,单倍型H_9和Hap_24/25/35/48是同一批样本,在2个系统树上均形成一个单独分支(图3),表明这些单倍型是长江上游特有单倍型,适应了上游生态环境,宜昌的一尾样本很可能来自上游群体。以鱼类线粒体cytb基因0.65%的突变速率[23]和控制区3.6%的突变速率[24]为基准,估算2个谱系间的分化时钟,其分化时间为距今45 536(Cytb)-49 411(D-loop)年前,依此推测2个谱系的分化时间大约在第四纪更新世晚期,这期间温差有12 ℃左右,推测是在地貌变化和种群演化两方面因素影响下形成,可能是冰期和间冰期交替导致地理隔离与融合造成单倍型分化,使得物种形成了不同种群,种群繁衍至今有了现在的遗传结构模式。
遗传多样性的本质是遗传变异,是生命进化和物种分化的基础,生物的遗传变异有一个积累的过程,当积累到一定程度,这些变异可能对该物种的遗传结构、种群分化甚至是物种的进化产生影响[25]。本研究结果显示,长江上游和中游群体存在显著遗传分化,上游群体遗传多样性高于中游群体,这对以后的渔业管理工作提供了一定的理论基础,例如增殖放流要谨遵“哪里来哪里放”原则,在上游增殖放流应选用上游原种种苗,中游的只能选用中游群体原种,避免跨流域种苗放流可能带来的潜在生态风险。
[1]李思发,蔡正纬,陆伟民,等.长江水系鲢鱼和珠江水系鲢鱼的生长差异[J].水产学报,1984,(3):211-218.
[2]田辉伍,何 春,刘明典,等.长江上游干流三层流刺网渔获物结构研究[J].淡水渔业,2016,46(5):37-42.
[3]刘绍平,陈大庆,段辛斌,等.长江中上游四大家鱼资源监测与渔业管理[J].长江流域资源与环境,2004,13(2):183-186.
[4]李小芳.鲢(Hypophthalmichthysmolitrix)亲本增殖放流遗传效果评估[D].重庆:西南大学,2012.
[5]Gonzalez E B,Aritaki M,Knutsen H,et al.Effects of large-scale releases on the genetic structure of red sea bream(Pagrusmajor,Temminck et Schlegel)Populations in Japan[J].Plos One,2015,10(5):e0125743.
[6]Lorenzen K,Beveridge M C M,Mangel M.Cultured fish:integrative biology and management of domestication and interactions with wild fish[J].Biol Rev,2012,87(3):639-660.
[7]Zhao L,Chenoweth E L,Liu J,et al.Effects of dam structures on genetic diversity of freshwater fishSinibramamacrops,in Min River,China[J].Biochem Syst Ecol,2016,68:216-222.
[8]Ardren W R,Bernall S R.Dams impact westslope cutthroat trout metapopulation structure and hybridization dynamics[J].Conserv Genet,2017,18(2):297-312.
[9]张四明,汪登强,邓 怀,等.长江中游水系鲢和草鱼群体mtDNA遗传变异的研究[J].水生生物学报,2002,26(2):142-147.
[10]于 悦,庞美霞,俞小牧,等.利用微卫星分子标记分析长江、赣江和鄱阳湖鲢群体遗传结构[J].华中农业大学学报,2016,35(6):104-110.
[11]Li S F,Xu J W,Yang Q L,et al.Significant genetic differentiation between native and introduced silver carp(Hypophthalmichthysmolitrix)inferred from mtDNA analysis[J].Environ Biol Fishes,2011,92(4):503-511.
[12]王长忠,梁宏伟,邹桂伟,等.长江中上游两个鲢群体遗传变异的微卫星分析[J].遗传,2008,30(10):1341-1348.
[13]庞美霞,俞小牧,童金苟.三峡库区5个鲢群体遗传变异的微卫星分析[J].水生生物学报,2015,39(5):869-876.
[14]Kocher T D,Thomas W K,Meyer A,et al.Dynamics of mitochondrial DNA evolution in animals:amplification and sequencing with conserved primers[J].Proc Natl Acad Sci United States of America,1989,86(16):196-200.
[15]Okazaki T.Genetic relationships of Japanese and Korean Bagrid catfishes inferred from mitochondrial DNA analysis[J].Zool Sci,1999,16:363-379.
[16]陈会娟,汪登强,段辛斌,等.长江中游鳊群体的遗传多样性[J].生态学杂志,2016,35(8):2175-2181.
[17]Thompson J D,Gibson T J,Plewniak F,et al.The CLUSTALX windows interface;flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Res,1997,(2):4876-4882.
[18]Tamura K,Stecher G,Peterson D,et al.MEGA6:Molecular evolutionary genetics analysis Version 6.0[J].Mol Biol Evol,2013,30:2725-2729.
[19]Rozas J,Rozas R.DNASP Version 3:an integrated program for molecular population genetics and molecular evolution analysis[J].Bioinformatics,1999,15:174-175.
[20]Excoffier L,Smouse P E,Quattro J M.Analysis of molecular variance inferred from metric distances among DNA haplotypes:application to human mitochondrial DNA restriction data[J].Genetics,1992,131:479-491.
[21]陈文静,张燕萍,段辛斌,等.基于线粒体控制区全序列的鄱阳湖水系鲢增殖放流群体与野生群体的遗传多样性分析[J].中国农学通报,2013,29(35):89-95.
[22]Katamachi D,Ikeda M,Dong S,et al.Genetic variability of silver carpHypophthalmichthysmolitrixpopulation introduced into the Tone River basin,Japan assessed by mtDNA analysis[J].Nihon-suisan-gakkai-shi,2011,77(1):31-39.
[23]Zardoya R,Doadrio I.Molecular evidence on the evolutionary and biogeographical patterns of European cyprinids[J].J Mol Evol,1999,49:227-237.
[24]Donaldson K A,Wilson R R.Amphi-Panamic geminates of snook(Percoidei:Centropomidae)provide a calibration of the divergence rate in the mitochondrial DNA control region of fishes[J].Mol Phylogen Evol,1999,13:208-213.
[25]陈宜喻.淡水生态系统中的若干生物多样性问题[J].生物科学信息,1990,2(5):197-200.
声明:本站属公益性没有商业目的的网站,上列文章仅供个人学习参考。本站所发布文章为原创的均标注作者或来源,未经授权不得转载,许可转载的请注明出处。本站所载文章除原创外均来源于网络,如有未注明出处或标注错误或侵犯了您的合法权益,请及时联系我们!
欢迎关注本站(可搜索)"养鱼E线"微信公众帐号和微信视频号"养鱼一线"以及头条号"水花鱼@渔人刘文俊"!

